CD56


Cette section ou cet article est une traduction incomplète ().
Vous pouvez modifier la page pour effectuer la traduction.
| NCAM1 | |||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
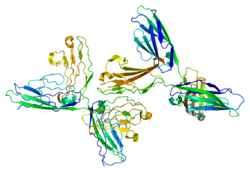 | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Structure de la protéine NCAM1. Basé sur l'identifiant PDB 1epf. | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Identifiants | |||||||||||||||||||||||||||||||||||||||||||||||||||
| Aliases | NCAM1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| IDs externes | OMIM: 116930 MGI: 97281 HomoloGene: 40754 GeneCards: NCAM1 | ||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
| Wikidata | |||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||
L'antigène CD56 (cluster de différenciation 56) ou NCAM (Neural Cell Adhesion Molecule) est une glycoprotéine s'exprimant à la surface des neurones, des glia et des muscles du squelette. Le CD56 a été impliqué dans l'adhésion des cellules entre elles, le développement des neurites, la plasticité des synapses, l'apprentissage et la mémoire.
D'autre part, le CD56 est l'antigène de surface qui caractérise les cellules Natural Killer (NK cells en anglais) humaines qui sont des lymphocytes du système immunitaire inné. Cet antigène n'est pas présent sur les cellules NK murines et aucun autre antigène approchant n'a été décrit.

Texte anglais à traduire :
There are at least 27 alternatively spliced NCAM mRNAs produced giving a wide diversity of NCAM isoforms. The three main isoforms of NCAM vary only in their cytoplasmic domain: NCAM-120kDa (GPI anchored), NCAM-140kDa (short cytoplasmic domain), NCAM-180kDa (long cytoplasmic domain).
The extracellular domain of NCAM consists of five immunoglobulin-like (Ig) domains followed by two fibronectin type III (FNIII) domains. The different domains of NCAM have been shown to have different roles with the Ig domains being involved in homophilic binding to NCAM, and the FNIII domains being involved signalling leading to neurite outgrowth. Homophilic binding occurs between NCAM molecules on opposing surfaces (trans-) and NCAM molecules on the same surface (cis-)1. There is much controversy as to how exactly NCAM homophilic binding is arranged both in trans- and cis-. Current models suggest trans- homophilic binding occurs between two NCAM molecules binding antiparallel between all five Ig domains or just IgI and IgII. cis- homophilic binding is thought to occur by interactions between both IgI and IgII, and IgI and IgIII, forming a higher order NCAM multimer. Both cis- and trans- NCAM homophilic binding have been shown to be important in NCAM “activation” leading to neurite outgrowth.
Another layer of complexity is created by the insertion of other "minor" exons in the NCAM transcript. The two most notable are the VASE (VAriable domain Spliced Exon)exon which is thought to correlate with an inbition of the neurite outgrowth promoting properties of NCAM, and the MSD (Muscle Specific Domain) the function of which is unknown but is found only in NCAM-120 in skeletal mucle.
NCAM can be posttranlationally modified by the addition of polysialic acid to the fifth Ig domain which is thought to abrogate its homophilic binding properties and can lead to reduced cell adhesion important in cell migration and invasion.
NCAM is thought to signal to induce neurite outgrowth via the Fibroblast Growth Factor Receptor (FGFR) and act upon the p59Fyn signalling pathway.
In anatomic pathology, pathologists make use of CD56 immunohistochemistry to recognize certain tumors. Normal cells that stain positively for CD56 include NK cells, activated T cells, the brain and cerebellum, and neuroendocrine tissues. Tumors that are CD56-positive are myeloma, myeloid leukemia, neuroendocrine tumors, Wilm's tumor, adult neuroblastoma, NK/T cell lymphomas, pancreatic acinar cell carcinoma, pheochromocytoma, and small cell lung carcinoma.
Ewing's sarcoma / PNET is CD56 negative.
Lien externe
- Pathology Outlines
v · m Liste de clusters de différenciation | |
|---|---|
| |
| Liste de molécules CD humaines |
 Portail de la biologie cellulaire et moléculaire
Portail de la biologie cellulaire et moléculaire  Portail de la médecine
Portail de la médecine
- ↑ a b et c GRCh38: Ensembl release 89: ENSG00000149294 - Ensembl, May 2017
- ↑ a b et c GRCm38: Ensembl release 89: ENSMUSG00000039542 - Ensembl, May 2017
- ↑ « Publications PubMed pour l'Homme », sur National Center for Biotechnology Information, U.S. National Library of Medicine
- ↑ « Publications PubMed pour la Souris », sur National Center for Biotechnology Information, U.S. National Library of Medicine


















